In der Ära der gezielten Therapie, Mutation Profilierung von Krebs ist ein entscheidender Aspekt therapeutische Entscheidungen zu treffen. Zu Krebs auf molekularer Ebene, die Verwendung von Formalin fixierten Paraffin eingebettetem Gewebe ist wichtig, zu charakterisieren. Wir testeten das Ion AmpliSeq Cancer Hotspot-Panel v2 und nCounter Copy Number Variation Assay in 89 Formalin-fixierten Magenkrebs Proben in Paraffin eingebettet, um zu bestimmen, ob sie in Archiv klinischen Proben für personalisierte zielgerichtete Therapien anwendbar sind. Wir validiert die Ergebnisse mit Sanger-Sequenzierung, quantitative Echtzeit-PCR, Fluoreszenz in situ-Hybridisierung und Immunhistochemie. Häufig entdeckt somatische Mutationen enthalten TP53 Citation:. Kim S, Lee J, Hong ME, Do IG, Kang SY, Ha SY, et al. (2014) Hochdurchsatz-Sequenzierung und Copy Number Variation Erkennung Mit Formalin fixiert eingebettete Gewebe beim metastasierten Magenkrebs. PLoS ONE 9 (11): e111693. doi: 10.1371 /journal.pone.0111693 Editor: Hiromu Suzuki, Sapporo Medical University, Japan Empfangen: 28. April 2014; Akzeptiert: 29. September 2014; Veröffentlicht: 5. November 2014 Copyright: © 2014 Kim et al. Dies ist eine Open-Access-Artikel unter den Bedingungen der Lizenz Creative Commons, die uneingeschränkte Nutzung erlaubt, die Verteilung und Vervielfältigung in jedem Medium, vorausgesetzt, der ursprüngliche Autor und Quelle genannt werden Datenverfügbarkeit. Die Autoren bestätigen, dass alle Daten, die Ergebnisse sind in vollem Umfang zur Verfügung, ohne Einschränkung zu Grunde liegen. Alle relevanten Daten sind in der Papier und seine Hintergrundinformationen Dateien Finanzierung:. Diese Studie wurde durch einen Zuschuss aus dem Korea Healthcare Technology R &unterstützt wurde; D Projekt, Ministerium für Gesundheit & Welfare Angelegenheiten, Republik Korea (A101130) und einem Samsung Biomedical Research Institute Zuschuss (# SBRI-SP1B20112). Diese Studie wurde auch von einem Samsung Medical Center intra-Zuschuss unterstützt, 20 x 20 Projekt (# GFO1140111). Co-Autoren Seokhwi Kim, Jeeyun Lee, Min Eui Hong, In-Gu Do, So Young Kang, Sang Yun Ha, Seung Tae Kim, Se Hoon Park, Won Ki Kang, Min-Gew Choi Jun Ho Lee, Tae Sung Sohn Jae Mond Bae, Sung Kim und Kyoung-Mee Kim werden von Samsung Medical Center eingesetzt. Co-Autor Duk-Hwan Kim wird von Samsung Biomedical Research Institute beschäftigt. Samsung Biomedical Research Institute und Samsung Medical Center zur Verfügung gestellt Unterstützung in Form von Gehältern für Autoren Seokhwi Kim, JL, MEH, IGD, SYK, SJH, STK, SHP, WKK, MGC, JHL, TSS, JMB, Sung Kim, KMK und DHK , aber im Studiendesign, Datenerhebung und Analyse haben keine zusätzliche Rolle, Entscheidung zu veröffentlichen oder um die Herstellung des Manuskripts nicht. Die spezifischen Rollen dieser Autoren sind in der 'Autor Beiträge Abschnitt artikuliert Konkurrierende Interessen:. Die Autoren haben die folgenden Interessen: Diese Studie wurde teilweise von Samsung Biomedical Research Institute und Samsung Medical Center finanziert. Co-Autoren Seokhwi Kim, Jeeyun Lee, Min Eui Hong, In-Gu Do, So Young Kang, Sang Yun Ha, Seung Tae Kim, Se Hoon Park, Won Ki Kang, Min-Gew Choi Jun Ho Lee, Tae Sung Sohn Jae Mond Bae, Sung Kim und Kyoung-Mee Kim werden von Samsung Medical Center eingesetzt. Co-Autor Duk-Hwan Kim wird von Samsung Biomedical Research Institute beschäftigt. Es gibt keine Patente, Produkte in der Entwicklung oder in Verkehr gebrachten Produkten zu erklären. Dabei werden nicht die Einhaltung der Autoren ändern, um alle Politiken PLoS ONE auf den Austausch von Daten und Materialien. Während Magenkrebs die vierthäufigste Krebs in der Welt ist, ist es die zweite Haupttodesursache. [1] Die Inzidenz ist in den asiatischen Ländern deutlich höher, einschließlich Korea, wo es der zweithäufigste Krebs. [2] Vor kurzem mehrere gezielte Therapeutika für Magenkrebs entdeckt worden, die für Ärzte zusätzlichen Optionen und Patienten [3] - [5] In der Ära der gezielten Therapie, Mutation Profilierung des verursachenden Krebs. ist für therapeutische Entscheidungen von entscheidender Bedeutung. Versuche Mutationen zum Profil wurden mit traditionellen Sanger-Sequenzierung gemacht; es ist jedoch keine optimale Verfahren in klinischen Umgebungen aufgrund der Kosten, Zeit und Arbeit erforderlich. Außerdem erfordert Sanger Sequenzierung erhebliche Mengen an DNA; geringe Mengen an Probe für mehrere Gene in der gleichen Zeit die Bewertung nicht möglich ist. Einführung von Next Generation Sequencing (NGS) Methoden hat beschlossen, dieses Problem durch die Multiplex-Hochdurchsatz-Sequenzierung vieler Proben für mehrere Gene gleichzeitig. [6], [7] Eine der NGS-Plattformen setzt das Ion Torrent AmpliSeq Cancer Feld auf nichtoptische Detektion von Wasserstoffionen in einer Halbleitervorrichtung, [8] und in der Lage, 2855 onkogene Mutationen in 50 allgemein mutierten Gene zu detektieren (Tabelle S1). Es ist besser als bei anderen Massenspektroskopie-basierten Sequenzierungsverfahren, Sequenzierung Bereitstellung Ergebnisse schneller und zu geringeren Kosten. [8] Sie ist anwendbar in Paraffin eingebettetem (FFPE) Gewebeproben mit geringen Mengen an DNA in Formalin fixiert. Weil es eine hohe Empfindlichkeit in Screening bekannt onkogenen Mutationen stellt sicher, [9], [10] das Ion Torrent AmpliSeq Krebs-Panel ist die Wahl der 5 großen Krebszentren in den Vereinigten Staaten für die molekulare Diagnostik in gezielte Therapie [11]. Die Amplifikation von Onkogenen ist ein wichtiger Mechanismus für die Gen-Überexpression und trägt zur Entwicklung von Tumoren. [12] Beispiele hierfür sind Amplifikation von HER2 Bessere zugeschnittene Krebstherapie kann das Outcome der Patienten zu verbessern. Patiententumorproben werden erforderlich sein, um Krebs auf molekularer Ebene zu charakterisieren und die Krankheit Untergruppen zu identifizieren, die verschiedene Behandlungen erhalten sollen. Die Verwendung von FFPE Gewebe ist wichtig für solche Studien zu ermöglichen. [16] wir AmpliSeq und nCounter benutzerdefinierte CNV Platten in FFPE Magenkrebs Proben hier getestet, um festzustellen, ob sie in Archiv klinischen Proben für personalisierte zielgerichtete Therapien anwendbar sind. Proben Tumorzellanteil mit mehr als 75% unter dem Mikroskop von 4 mm ungefärbten Abschnitten durch Vergleich mit einem H &seziert wurden; E gefärbten Objektträger, und genomische DNA wurde unter Verwendung eines Qiagen DNA FFPE Tissue Kit (Qiagen, Hilden, Deutschland) extrahiert, gemäß den Anweisungen des Herstellers von 96 Patienten mit fortgeschrittenem Magenkrebs. Nach der Extraktion gemessen wir Konzentration sowie 260/280 und 260/230 nm-Verhältnis von Spektrophotometer (ND1000, Nanodrop Technologies, Thermofisher Scientific, MA, USA). Jede Probe wurde dann mit dem Qubit Fluorometer (Life Technologies, Carlsbad, Kalifornien) quantifiziert. Genomische DNA mit > 10 von Qubit Fluorometer gemessen ng wurde Bibliothek Vorbereitung unterzogen und sieben Proben gescheitert Bibliotheken zu konstruieren und wurden von dieser Studie ausgeschlossen. Schließlich wurden 89 Fälle schließlich analysiert und enthalten 31 weibliche und 58 männliche Patienten. Tabelle 1 zeigt die klinischen und pathologischen Merkmale der Patienten in dieser Studie. Rezidive oder Metastasen mit einer medianen Nachbeobachtungszeit von 76 Monaten 11 Patienten entwickelt (Bereich 5,5 bis 149,3). Die Studie wurde von der Institutional Review Board (IRB) im Samsung Medical Center genehmigt. Alle klinischen Untersuchung wurde nach den Grundsätzen der Erklärung von Helsinki zum Ausdruck durchgeführt. Die schriftliche Einwilligung wurde aufgrund der retrospektiven Analyse durch das IRB verzichtet und anonyme Daten. Die Proben wurden im Rahmen einer medizinischen Routineverfahren gesammelt und wurden von den Autoren dieser Studie gesammelt. Proben von verstorbenen Patienten oder Live-Patienten wurden alle deidentifiziert, einschließlich der Entfernung von irgendwelchen und alle demografische Informationen, vor der Analyse und Einwilligungserklärung durch das IRB verzichtet. Wir haben das Ion AmpliSeq Krebs-Panel v2 (Ion Torrent) häufige somatische Mutationen zu erkennen, die auf Basis von Literatur ausgewählt wurden. Es wird untersucht 2855 Mutationen in 50 häufigsten mutierte Onkogene und Tumorsuppressorgene (Tabelle S1). Zunächst wurden 10 ng der DNA aus jedem der 89 FFPE Tumorproben unterzogen Einrohr-Multiplex-PCR-Amplifikation des Primer Ion AmpliSeqCancer Pool und die Ionen AmpliSeqKit Reagenzien (Life Technologies) verwendet. Behandlung des resultierenden Amplicons mit FUPA Reagent verdaut teilweise die Primer und phosphoryliert die Amplicons. Die phosphorylierten Amplikons wurden Ion-Adapter und gereinigt ligiert. Für barcodierte Bibliothek Vorbereitung substituierten wir barcodierte Adapter aus dem Ion Xpress Barcode-Adapter 1-96 Kit für den nicht-barcodierte Adapter-Mix in der Kit AmpliSeq Bibliothek Ion versorgt. Die ligierte DNA unterzog Nick-Translation und Verstärkung der Verbindung zwischen Adapter und Amplicons zu vervollständigen und ausreichend Material für die nachfolgende Vorlage Vorbereitung zu erzeugen. Zwei Runden Agencourt AMPure XP Bindungsreagens bei 0,6 und 1,2 bead-to-sample Volumenverhältnisse entfernt Eingangs DNA und nicht eingebauten Primer aus der Amplikons. Die endgültigen Bibliothek Moleküle waren 125~300 bp groß. Wir übertragen dann die Bibliotheken in das Ion OneTouch System für die automatisierte Vorlage Vorbereitung. Die Sequenzierung wurde auf dem Ion PGM-Sequenzer durchgeführt gemäß den Anweisungen des Herstellers. Wir haben IonTorrent Software für die automatisierte Datenanalyse. die Sensitivität und Spezifität des Ion AmpliSeq Krebs-Panel zu messen, ganze Exoms Sequenzierungsergebnisse aus 4 Magenkrebs Proben mit bekannten Mutationsstatus verwendet wurden [17]. nCounter Copy Number Variation Codesätzen Für den Nachweis von CNV, nCounter Copy Number Variation Codesets wurden mit 300 ng gereinigte genomische DNA von 2 bis 3 Abschnitte von 4 um dicke FFPE repräsentative Tumorblöcke mit QIAamp extrahiert verwendet DNA FFPE Tissue Kit (Qiagen, Hilden, Deutschland). DNA wurde durch AluI-Verdau fragmentiert und bei 95 ° C denaturiert. Fragmentierte DNA wurde in dem nCounter Cancer CN Assay Kit (nano Technologies) bei 65 ° C für 18 Stunden mit dem codeset von 86 Genen hybridisiert und verarbeitet gemäß den Anweisungen des Herstellers. Der nCounter Digital-Analyzer gezählt und tabellarisch die Signale von Reportersonden und mittlere Zählwerte von > 3 wurden durch IHC, FISH oder Echtzeit-PCR genannt und bestätigt IHC für HER2, EGFR (HER 1) und CCNE1. für die Validierung von CNV Ergebnisse von nCounter erhalten, führten wir IHC für HER2 in allen Fällen und EGFR und CCNE1 in ausgewählten Fällen. Nach Entparaffinierung und Rehydrierung, 4 mm Abschnitte auf Silan-beschichtete Objektträger wurden für HER2 immunhistochemisch. Die HercepTest (Dako, Glostrup, Dänemark) wurde gemäß den Richtlinien des Herstellers verwendet, wie zuvor beschrieben. [18] Für EGFR haben wir anti-NCL-L-EGFR-384 Maus monoklonalen primären Antikörper (1:100 Verdünnung; Novocastra /Vision Biosystems, Newcastle, UK) und für CCNE1 wir verwendeten anti-CCNE1 /Cyclin E1-Antikörper (Klon HE12; 1:200 Verdünnung; Thermo Fisher Scientific, MA). Die XT Ventana BenchMark automatisierten Objektträger-Verarbeitungssystem wurde gemäß dem Protokoll des Herstellers verwendet. Ein Experte Pathologen (KMK) bewertet die Ergebnisse FISH für HER2 FISH wurde DNA-spezifische Sonden aus PathVision ™ (Abbott /Vysis mit Dual-Farbe durchgeführt. LSI HER2 SpectrumOrange ™ und CEP 17 SpectrumGreen ™), wie zuvor in Fällen mit zweideutigen HER2 Überexpression beschrieben. [19] Wir zählten die Hybridisierungssignale in 20 Kerne pro Probe unter einem Fluoreszenzmikroskop (Zeiss Axioskop) unter Verwendung von Filtersätzen von Vysis (DAPI /Spectrum orange dual Bandpaß, DAPI /Spectrum Grün dual Bandpaß) empfohlen. Alle überlappenden Kerne wurden ausgeschlossen, und nur Kerne mit einer deutlichen Kerngrenze wurden ausgewertet. HER2 Wir DNAs von FFPE Magenkarzinom Tumorgewebe erhalten wird, verwendet. Die Reaktionsmischung enthielt 2 &mgr; l genomische DNA-Matrize, 10 uL von Taqman PCR Master universal Mischung (Applied Biosystems Inc., Foster City, CA) und 0,2 uM jedes Primers. Für eine genaue Erfassung der KN-Veränderungen, analysierten wir drei verschiedenen Regionen der KRAS Wir maßen Kopienzahl Verstärkung mit dem folgenden Profil: 2 min bei 50 ° C, Denaturierung bei 95 ° C von 40 Zyklen von 95 ° C für 15 sec und 60 ° C für 1 min, gefolgt 10 min. Wir stellten fest, relative Quantifizierung in vierfacher Ausfertigung, die 7900 HT schnelle Echtzeit-PCR-System. Ein RNaseP-Assay-Kit (Applied Biosystems) wurde als Kontrolle verwendet. Nach der Amplifikation eingeführt wir die Versuchsergebnisse Werte Schwellenwert-Zyklus für die Kopienzahl und Referenzassay in die CopyCaller Software (Applied Biosystems) für die post-PCR Datenanalyse enthält, wie zuvor beschrieben. [22] Wir ordneten die CN Verstärkung Status und die Anzahl der KRAS Wir schlossen alle auch Veränderungen nach einer automatisierten Mutation-Aufruf Algorithmus vermutlichen Mutationen zu erkennen verwendet wurde. Wiederholte Anrufe in mehr als 10 von 89 Proben wurden als falsch positiv angesehen und wurden ausgeschlossen. Wir verwendeten Cutoff-Werte von mehr als 6% Variante Frequenz und mehr als X100 Abdeckung wahr Mutationsänderungen in Übereinstimmung mit früheren Studien und eigener Erfahrung zu erkennen. [9], [10] gefiltert Wir Einzelnukleotid-Polymorphismen nach manueller Überprüfung der einzelnen Polymorphismus im Katalog der somatische Mutationen in Krebs (COSMIC, http://cancer.sanger.ac.uk/cancergenome/projects/cosmic) aus ( Abbildung 1). Für bekannte Gene in Magen-Karzinome mutiert ( TP53 Ergebnisse
(28,17%), APC
(10,1%), PIK3CA
(5,6%), KRAS
(4,5 %), SMO
(3,4%), STK11
(3,4%), CDKN2A
(3,4%) und SMAD4
(3,4%) . Vergrösserungen von HER2
, CCNE1
, MYC
, KRAS
und Gene
EGFR wurden in 8 (8,9%) beobachtet , 4 (4,5%), 2 (2,2%), 1 (1,1%) und 1 (1,1%) Fälle, respectively. In den Fällen mit der Amplifikation Fluoreszenz-in situ-Hybridisierung für HER2
verifiziert Genamplifikation und Immunhistochemie für HER2, EGFR und CCNE1 prüft die Überexpression von Proteinen in Tumorzellen. Abschließend wir auf Halbleiterbasis Sequenzierung und nCounter Kopienzahl Variation Analysen in Formalin-fixierten und in Paraffin eingebetteten Magenkrebs Proben erfolgreich durchgeführt. Hochdurchsatz-Screening in Archivierung klinischer Proben ermöglicht eine schnellere, genauere und kosteneffiziente Erfassung von Hotspot-Mutationen oder Verstärkung in den Genen
Einführung
, MET
, FGFR2-
und KRAS
Gene in Magenkrebs. [13], [14] In den Nachweis von Variationen der Kopienzahl (CNV) in klinischen Proben, Fluoreszenz in situ Hybridisierung (FISH) und /oder Immunhistochemie (IHC) weithin verwendet worden ist. Doch hohe Kosten und kleine Probengrößen von Biopsiematerial begrenzen die Anwendung dieser Methoden, und es gibt immer noch ein Bedarf an weiteren Hochdurchsatz-Technologie mit einfacher Zugänglichkeit, hohe Empfindlichkeit und niedrige Kosten. nCounter CNV-Codesätzen (nano Technologien, Life Sciences, Seattle, WA) bieten hohe Genauigkeit und Reproduzierbarkeit von Studien aller Größen und produzieren bessere, schnellere Ergebnisse mit wesentlich weniger Aufwand als bei quantitative Echtzeit-Polymerase-Kettenreaktion (qPCR) oder CNV-Arrays [ ,,,0],15].
Materialien und Methoden
Ion AmpliSeq Krebs Panel v2
Gen amplifiziert wurde betrachtet, wenn die FISH-Signal-Verhältnis von HER2 /CEP17
war größer als oder gleich 2,0 [20].
Real-time PCR für KRAS und MET Verstärkung
Gen: eine Region innerhalb des Introns 1 (TaqMan Copy Number Assay Hs06943812_cn), einen Bereich innerhalb des Introns 2 (Hs002534878_cn) und einen Bereich innerhalb von Exon 6 (Hs02739788_cn). . Für MET
Gen, wir die Primer verwendet, wie zuvor beschrieben [21]
Kopien basierend auf der Übereinstimmung der Ergebnisse in mindestens zwei der drei Sonden.
Analytische Methoden
, APC
, PIK3CA
, STK11
, CDKN2A
, KRAS
, HRAS
, BRAF
und CTNNB1
), eine manuelle Überprüfung von automatischen Anruf Ergebnissen durchgeführt wurde schädliche Mutationen zu fangen mit leicht low- Variante Frequenz.
 PENTAX Medical sammelt $125,
PENTAX Medical sammelt $125,
 Neil Bell zum Chief Development Officer von Avacta Life Sciences ernannt
Neil Bell zum Chief Development Officer von Avacta Life Sciences ernannt
 Mikrobiom könnte bei Frauen mit HPV . helfen, das Risiko einer Krebsvorstufe zu erkennen
Mikrobiom könnte bei Frauen mit HPV . helfen, das Risiko einer Krebsvorstufe zu erkennen
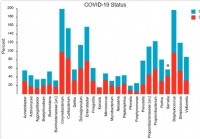 Zusammensetzung und Struktur des nasopharyngealen Mikrobioms beziehen sich auf die Schwere der COVID-19-Krankheit
Zusammensetzung und Struktur des nasopharyngealen Mikrobioms beziehen sich auf die Schwere der COVID-19-Krankheit
 Stuhltransplantation von bestimmten Spendern besser als von anderen
Stuhltransplantation von bestimmten Spendern besser als von anderen
 Führt Bluthochdruck immer zu schwerem COVID-19?
Führt Bluthochdruck immer zu schwerem COVID-19?
 Menschliches Mikrobiom trimmt Schleimhautglykane,
Beeinflussung der SARS-CoV-2-Infektion Ein internationales Forscherteam hat eine Studie durchgeführt, die zeigt, dass Unterschiede im menschlichen Mikrobiom die Fähigkeit des schweren akuten respirato
Menschliches Mikrobiom trimmt Schleimhautglykane,
Beeinflussung der SARS-CoV-2-Infektion Ein internationales Forscherteam hat eine Studie durchgeführt, die zeigt, dass Unterschiede im menschlichen Mikrobiom die Fähigkeit des schweren akuten respirato
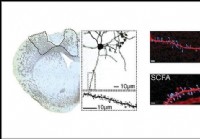 Die Nahrungsergänzung mit kurzkettigen Fettsäuren verbessert die Erholung von Schlaganfällen,
Mäusestudie zeigt Die Ergänzung der kurzkettigen Fettsäuren des Körpers kann die Erholung von Schlaganfällen verbessern. nach einer Studie an Mäusen, die kürzlich in JNeurosci veröffentlicht wurde. Di
Die Nahrungsergänzung mit kurzkettigen Fettsäuren verbessert die Erholung von Schlaganfällen,
Mäusestudie zeigt Die Ergänzung der kurzkettigen Fettsäuren des Körpers kann die Erholung von Schlaganfällen verbessern. nach einer Studie an Mäusen, die kürzlich in JNeurosci veröffentlicht wurde. Di
 Darmmikrobiom und CED – der Zusammenhang vielleicht in der Ernährung sagt Studie
Eine neue Studie hat schlüssige Beweise dafür erbracht, dass entzündliche Darmerkrankungen oder IBD eng mit der mikrobiellen Umgebung des Darms verbunden sind, die durch verschreibungspflichtige Diäte
Darmmikrobiom und CED – der Zusammenhang vielleicht in der Ernährung sagt Studie
Eine neue Studie hat schlüssige Beweise dafür erbracht, dass entzündliche Darmerkrankungen oder IBD eng mit der mikrobiellen Umgebung des Darms verbunden sind, die durch verschreibungspflichtige Diäte